Google Deepmind ha presentado una nueva versión AlphaFold, su modelo transformador de aprendizaje automático que predice la forma y el comportamiento de las proteínas. AlphaFold 3 no sólo es más preciso, sino que predice interacciones con otras biomoléculas, lo que la convierte en una herramienta de investigación mucho más versátil, y la compañía está poniendo a disposición en línea una versión limitada del modelo de forma gratuita.
Desde el debut del primer AlphaFold en 2018, el modelo sigue siendo el método líder para predecir la estructura de las proteínas a partir de la secuencia de aminoácidos que las componen.
Aunque esto parece una tarea bastante limitada, es fundamental para casi toda la biología comprender las proteínas, que realizan una variedad casi infinita de tareas en nuestros cuerpos, a nivel molecular. En los últimos años, las técnicas de modelado computacional como AlphaFold y RoseTTaFold han reemplazado a los costosos métodos de laboratorio, acelerando el trabajo de miles de investigadores en otros tantos campos.
Pero la tecnología todavía es un trabajo en progreso, y cada modelo es “sólo un paso en el camino”, como lo expresó el fundador de Deepmind, Demis Hassabis, en una conferencia de prensa sobre el nuevo sistema. La compañía adelantó el lanzamiento a fines del año pasado, pero este marca su debut oficial.
Dejaré que los blogs científicos expliquen exactamente cómo el nuevo modelo mejora los resultados, pero baste decir aquí que una variedad de mejoras y técnicas de modelado han hecho que AlphaFold 3 no sólo sea más preciso, sino también más aplicable.
Una de las limitaciones del modelado de proteínas es que incluso si sabes cómo se formará una secuencia de aminoácidos, eso no significa que necesariamente sepas a qué otras moléculas se unirá y cómo. Y si realmente queríamos hacer cosas con estas moléculas, como es lo que hace la mayoría, teníamos que descubrirlo mediante modelos y pruebas más laboriosos.
“La biología es un sistema dinámico y hay que entender cómo surgieron las propiedades de la biología a través de la interacciones entre diferentes moléculas de la célula. Y puedes pensar en AlphaFold 3 como nuestro primer gran paso hacia eso”, dijo Hassabis. “Es capaz de modelar proteínas que interactúan, por supuesto, con otras proteínas, pero también con otras biomoléculas, incluidas, sobre todo, cadenas de ADN y ARN”.
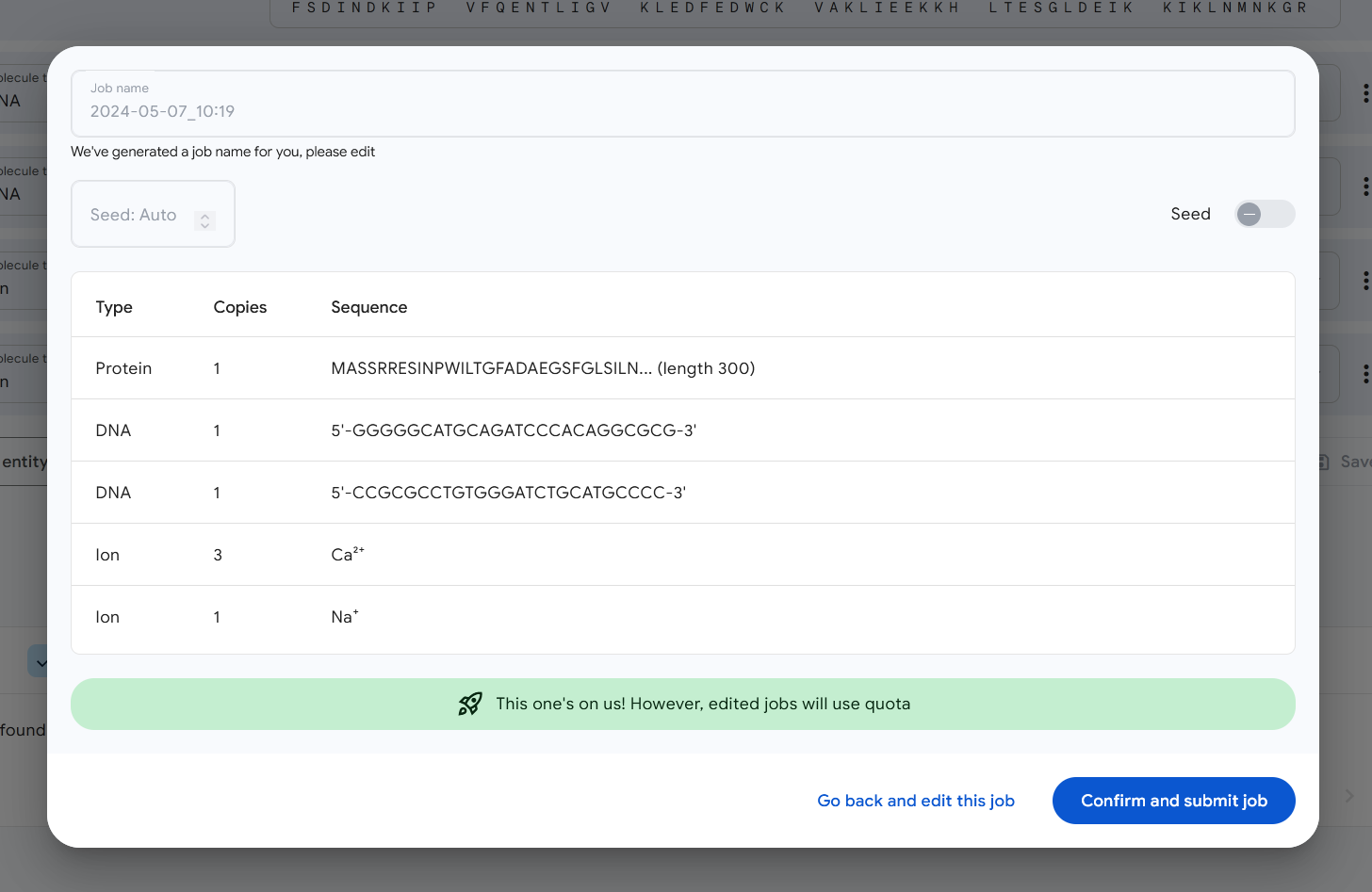
AlphaFold 3 permite simular múltiples moléculas a la vez, por ejemplo, una hebra de ADN, algunas moléculas de unión al ADN y tal vez algunos iones para darle vida. Esto es lo que se obtiene con una de esas combinaciones específicas, con las cintas de ADN subiendo por el medio, las proteínas brillando a un lado, y creo que esos son los iones anidados en el medio como pequeños huevos:
Esto, por supuesto, no es un descubrimiento científico en sí mismo. Pero incluso descubrir que una proteína experimental se uniría de algún modo, o de esta manera, o se contorsionaría hasta adoptar esta forma, era generalmente cuestión de días como mínimo o quizá de semanas o meses.
Si bien es difícil exagerar el entusiasmo en este campo en los últimos años, los investigadores se han visto paralizados en gran medida por la falta de modelos de interacción (de los cuales la nueva versión ofrece una forma) y la dificultad para implementar el modelo.
Este segundo problema es quizás el mayor de los dos, ya que si bien las nuevas técnicas de modelado eran “abiertas” en cierto sentido, al igual que otros modelos de IA, no son necesariamente fáciles de implementar y operar. Es por eso que Google Deepmind ofrece AlphaFold Server, una aplicación web gratuita y totalmente alojada que hace que el modelo esté disponible para uso no comercial.
Es gratis y bastante fácil de usar: lo hice en otra ventana de la llamada mientras lo explicaban (así es como obtuve la imagen de arriba). Solo necesita una cuenta de Google y luego le proporciona tantas secuencias y categorías como pueda manejar (se proporcionan algunos ejemplos) y la envía; en unos minutos su trabajo estará terminado y se le entregará una molécula 3D viva coloreada para representar la confianza del modelo en la conformación en esa posición. Como puede ver en el de arriba, las puntas de las cintas y aquellas partes más expuestas a átomos rebeldes son más claras o rojas para indicar menos confianza.
Pregunté si había alguna diferencia real entre el modelo disponible públicamente y el que se utiliza internamente; Hassabis dijo que “Hemos puesto a disposición la mayoría de las capacidades del nuevo modelo”, pero no dio más detalles.
Claramente es Google quien está haciendo todo lo posible, mientras que hasta cierto punto se queda con lo mejor, lo cual por supuesto es su prerrogativa. Crear una herramienta alojada y gratuita como esta implica dedicar recursos considerables a la tarea; no se equivoquen, esto es un pozo de dinero, una versión shareware costosa (para Google) para convencer a los investigadores del mundo de que AlphaFold 3 debería ser, al menos. Al menos, una flecha en su aljaba.
Créditos de imagen: Mente profunda de Google
Sin embargo, eso está bien, porque la tecnología probablemente imprimirá dinero a través de la subsidiaria de Alphabet (lo que la convierte en… ¿prima de Google?) Isomorphic Labs, que está utilizando herramientas computacionales como AlphaFold para trabajar en el diseño de fármacos. Bueno, hoy en día todo el mundo utiliza herramientas computacionales, pero Isomorphic fue el primero en utilizar los últimos modelos de Deepmind, combinándolos con “algunas cosas más patentadas relacionadas con el descubrimiento de fármacos”, como señaló Hassabis. La empresa ya tiene asociaciones con Eli Lilly y Novartis.
Sin embargo, AlphaFold no es el principio y el fin de la biología: solo una herramienta muy útil, como estarán de acuerdo innumerables investigadores. Y les permite hacer lo que Max Jaderberg de Isomorphic llamó “diseño racional de fármacos”.
“Si pensamos, día a día, cómo esto tiene un impacto en los laboratorios isomórficos: permite a nuestros científicos, a nuestros diseñadores de fármacos, crear y probar hipótesis a nivel atómico, y luego, en cuestión de segundos, producir predicciones estructurales altamente precisas… para ayudar. los científicos razonan sobre cuáles son las interacciones a realizar y cómo avanzar en esos diseños para crear un buen fármaco”, dijo. “Esto se compara con los meses o incluso años que podrían llevar hacerlo de forma experimental”.
Si bien muchos celebrarán el logro y la amplia disponibilidad de una herramienta alojada y gratuita como AlphaFold Server, otros pueden señalar con razón que esto no es realmente una victoria para la ciencia abierta.
Al igual que muchos modelos patentados de IA, el proceso de entrenamiento de AlphaFold y otra información crucial para replicarlo (una parte fundamental del método científico, como recordará) se ocultan en gran medida y cada vez más. Si bien el artículo publicado en Nature analiza con cierto detalle los métodos de su creación, faltan muchos detalles y datos importantes, lo que significa que los científicos que quieran utilizar la herramienta de biología molecular más poderosa del planeta tendrán que hacerlo bajo el atenta mirada de Alphabet, Google y Deepmind (quién sabe quién lleva realmente las riendas).
Los defensores de la ciencia abierta han dicho durante años que, si bien estos avances son notables, a la larga siempre es mejor compartir este tipo de cosas abiertamente. Después de todo, así es como avanza la ciencia y, de hecho, también cómo han evolucionado algunos de los programas informáticos más importantes del mundo.
Hacer que AlphaFold Server sea gratuito para cualquier aplicación académica o no comercial es, en muchos sentidos, un acto muy generoso. Pero la generosidad de Google rara vez viene sin condiciones. Sin duda, muchos investigadores aprovecharán este período de luna de miel para utilizar el modelo tanto como sea humanamente posible antes de que el resto caiga.

